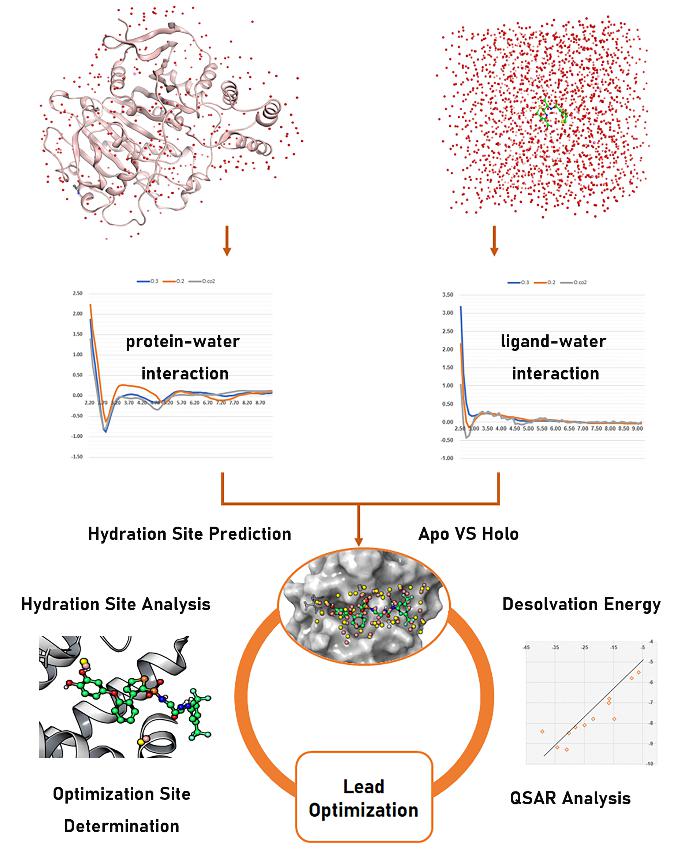
水分子在蛋白-配体结合中的重要性已为研究者们广泛接受。通过对关键水分子的分析可以帮助人们理解一些“反常”的结构-活性关系,指导先导化合物的优化设计。传统基于分子动力学模拟或蒙特卡洛模拟的方法虽然有较高的精度,但是计算非常耗时,且流程复杂,不易于实用化推广。
复旦大学药学院王任小课题组在前期工作中开发了一种经验方法HydraMap。该方法基于统计势预测蛋白结合口袋中的水合位点和计算去溶剂化能,因而在计算精度和速度之间实现合理平衡。近期,课题组对该方法做了重要改进,推出了加强版HydraMap v.2,相关工作以“HydraMap v.2: Prediction of Hydration Sites and Desolvation Energy with Refined Statistical Potentials”为封面文章发表于美国化学会旗下计算化学权威杂志J. Chem. Inf. Model。
|
|
图-1 JCIM杂志封面和HydraMap v.2工作流程图
该研究利用分子动力学模拟技术构建了近10000个有机小分子的溶剂化模型,并在此基础上推导出评价配体-水分子相互作用的统计势。同时,基于PDB(2020版)中17042个非冗余蛋白晶体结构更新了评估蛋白-水分子相互作用的统计势。基于这两组统计势,HydraMap v.2能够同时预测结合配体前后蛋白结合口袋中的水合位点分布变化,并识别出参与结合过程的关键水分子,如形成桥连氢键的保守水分子和可被取代的不稳定水分子。基于HydraMap v.2预测结果,本工作成功复现了一组MCL-1小分子抑制剂的合理优化过程。此外,基于结合配体前后的水合位点能量变化,该工作提出了计算去溶剂化能的新方法。在六个代表性靶标的测试集上,去溶剂化能计算值与实验亲合性数据表现出较好的相关性,很好地解释了同类骨架化合物的结构-活性关系。这些结果表明,HydraMap v.2为评估蛋白-配体结合过程中的去溶剂化效应提供了一种经济高效的解决方案,并可作为指导基于结构先导化合物优化的实用化工具。
复旦大学药学院李嫣副研究员为该论文的第一作者,复旦大学药学院王任小研究员为通讯作者。该研究得到了国家自然科学基金和国家重点研究计划的资助。
原文链接:https://pubs.acs.org/doi/full/10.1021/acs.jcim.3c00408(提供HydraMap v.2的可执行程序和说明文档,用户可免费下载使用。)