
寻找生物活性分子的潜在分子靶标(即反向钓靶,Target-Fishing)是现代药物发现中的关键环节,对于表型筛选后续的机制研究、天然产物分析、药物副作用预测以及老药新用都具有不可替代的作用。尽管亲和色谱等实验方法被广泛应用,但其耗时、昂贵且通量较低的局限性日益凸显。随着人工智能的发展,计算靶标预测作为一种高效的替代手段应运而生。然而现有方法往往局限于单一的算法逻辑:基于“相似分子具有相似活性”的配体假设;依赖蛋白质结构的对接评分;基于纯序列的单模态信息。由于药物-靶标相互作用数据的稀疏性以及单一算法的固有偏差,现有的计算工具在预测精度和结果解释性上仍面临巨大挑战,难以满足精准药物研发的需求。
近年来,复旦大学药学院王任小团队开发了一种名为COMET(Combined Matrix for Elucidating Targets)的反向钓靶预测算法。该研究构建了一个包含配体相似性评分、基于结构的结合亲和力预测以及分子对接的组合式框架,并通过机器学习算法实现了靶标的高精度排序。日前,该项工作以“COMET: A Machine-Learning Framework Integrating Ligand-Based and Target-Based Algorithms for Elucidating Drug Targets”为论文标题,在线发表在药物化学领域权威期刊Journal of Medicinal Chemistry上。
COMET框架的创新之处在于其“多元组合式”的架构设计(图1)。研究团队摒弃了传统的单一评分策略,而是构建了一个包含四大模块的综合系统。首先,配体模块采用改进的累积相似性分析、聚类关联算法和最大相似度算法,对查询分子与知识库中已知配体进行多层次比对,还通过统计学模型量化了相似性的显著程度。其次,靶标模块引入了课题组前期开发的蛋白-配体亲合性预测模型PLANET,在不进行构象采样的情况下快速预测分子与靶标结合口袋的结合强度。再次,COMET利用随机森林算法作为决策的核心模块,将上述评分整合成概率输出。为了增强结果的解释性,系统还集成了GPU加速的AutoDock Vina模块,能够为每个预测靶标生成与查询分子的结合模式。整个系统建立在一个精心构建的知识库之上,涵盖了2685个与人类疾病高度相关的蛋白质靶标及近100万对配体-靶标相互作用的实验数据。
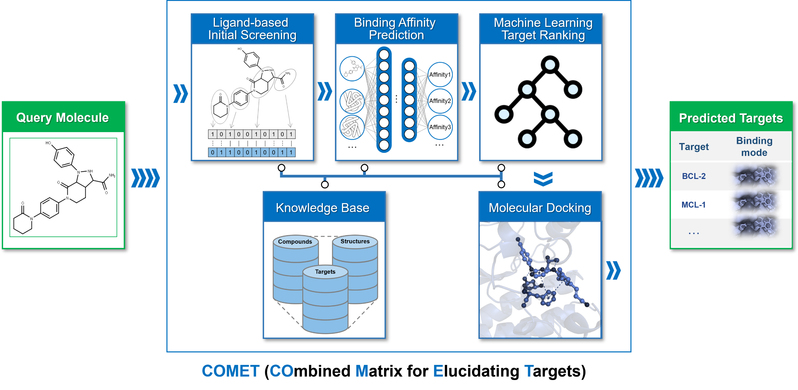
图1:COMET的模型框架示意图
为了客观评估模型性能,研究团队构建了两个包含500个多靶点配体的大型外部测试集(分别源自ChEMBL33和BindingDB),并与包括SwissTargetPrediction、SEA、PPB2、PharmMapper以及TransformerCPI 2.0在内的7种现有方法进行了横向对比。评估结果显示,COMET在各项关键指标上均展现出显著优势。在ChEMBL33测试集中,COMET的前100名召回率(Top 100 Recall)达到72.18%,前15名命中率(Top 15 Hit Rate)高达77.80%,远超排名第二的PPB2方法(分别为51.17%和63.80%)。在BindingDB测试集中,COMET的优势进一步扩大,前100名召回率和前15名命中率分别达到了78.75%和79.80%。值得注意的是,一些仅依赖蛋白序列的深度学习方法在此次基准测试中表现不佳,突显了在靶标预测中融合结构信息与配体先验知识的重要性。(图2)
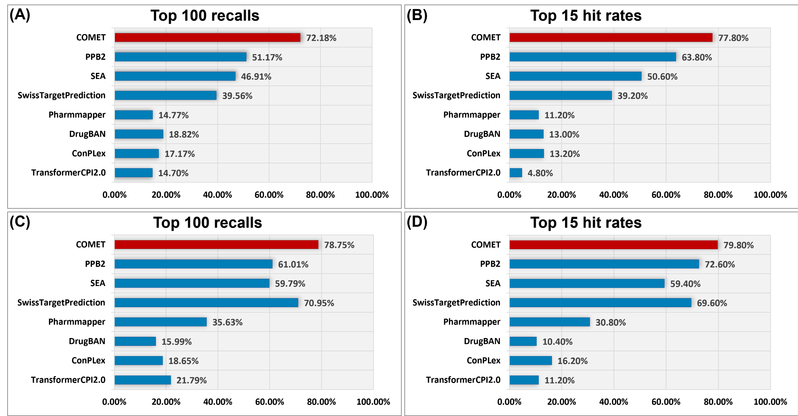
图2:COMET和其他7种靶点预测方法的性能对比结果。(A,B)在ChEMBL33测试集上的结果;(C,D)在BindingDB测试集上的结果。
COMET不仅在统计数据上表现优异,在应用中也展现了很高的实用性。得益于组合式的模型设计,COMET能够不仅输出概率分数,还为用户提供了预测的“证据链”。除了按概率排序的潜在靶点,COMET能为用户列出与查询分子结构相似的已知活性化合物,并展示预测的三维结合模式,以帮助用户根据先验知识进行判断和选择。以FDA批准的抗抑郁药物维拉佐酮(Vilazodone)为例,COMET成功在其预测列表的前12位中以超过90%的概率命中了所有已知靶标(包括5-HT1A受体等)。更重要的是,COMET预测出的第13至15位潜在靶标(多巴胺转运体、去甲肾上腺素转运体和hERG钾离子通道),虽然在系统知识库中未记录,但均被外部文献的实验数据所证实。COMET通过多层次的特征分析解释了预测依据:它识别出维拉佐酮含有与多巴胺转运体已知配体相似的取代吲哚骨架,并生成了合理的结合模式图,显示分子能精准占据DAT的中心结合口袋。这种从骨架相似性到结合构象合理性的全方位佐证,使COMET的预测结果具有很高的参考价值。(图3)
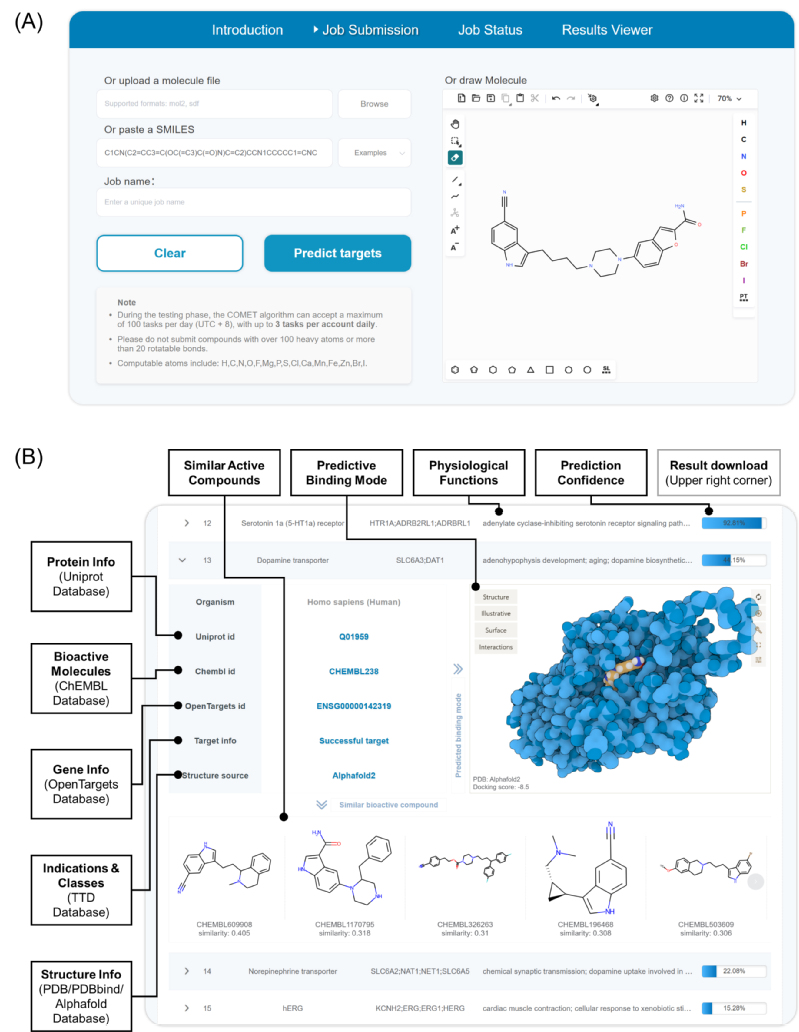
图3:以维拉佐酮作为查询分子的案例研究。(A)查询界面支持多种输入模式;(B)结果展示界面,包括多种基本信息、预测证据和基于分子对接的结合模式。
小结:该研究提出的COMET算法框架通过巧妙整合基于配体和基于靶标的算法,有效克服了单一策略的局限性,在保证计算速度的同时实现了预测精度的突破。该算法提供综合辅助证据的特性,也大大提升了其预测结果在药物研发实践中的价值。COMET算法已部署在该团队建设的PDBbind+网络服务器(https://www.pdbbind-plus.org.cn/comet)上供全球用户免费使用,自2024年上线以来已完成了超过2700次预测任务,为药物发现工作中的反向钓靶任务提供了新的强有力工具 。
复旦大学药学院博士生王浩杰为该论文第一作者。复旦大学药学院王任小研究员为该论文最后通讯作者。该工作得到国家自然科学基金重点项目、上海市科学技术委员会重点项目等资金支持。
原文链接:https://pubs.acs.org/doi/10.1021/acs.jmedchem.5c02652


